编者按:
尽管益生菌有着明确的定义,但是在食品和膳食补充剂中,益生菌一词却常常被误用和滥用。那么,究竟符合什么样标准的微生物才能够允许产品声明添加了“益生菌”呢?
今天我们特别翻译了由达能、科汉森、养乐多、杜邦等公司联合 ISAPP 执行科学官共同创作并发表在 Frontiers in Microbiology 杂志上关于食品和补充剂中益生菌标准的文章。希望该文能够为相关的产业人士和诸位读者带来一些帮助。
19 年后的今天,粮农组织/世卫组织(FAO/WHO)对益生菌的定义仍然具有很重要的意义,它可以被转化为四个简单而实用的标准,从而使人们得出结论,即特定的微生物菌株是否有资格作为食品和膳食补充剂中的益生菌使用。
益生菌菌株必须(i)充分被表征;(ii)预期用途具有安全性;(iii)至少根据公认的科学标准或根据当地/国家主管部门的建议和规定进行一项人体临床试验,并获得阳性结果;(iv)在产品的整个货架期内保持有效剂量。
我们提供了清晰和详细的信息,说明如何评估这四个标准。必须广泛采用这些标准,以确保在科学出版物、产品标签上以及与监管机构和公众的交流中正确使用益生菌一词。
以下是正文部分:
消费者对通过食物和膳食补充剂维持健康的兴趣日益浓厚。使用循证学方法来改善饮食和生活方式成为了一种持续增长的趋势。这产生了一个更加多样化的食品和补充剂市场,特别是那些含有益生菌的食品和补充剂。
2001 年,在世界卫生组织(WHO)和联合国粮食及农业组织(粮农组织)的组织下召开的专家咨询会议提出了一个实用的关于益生菌的定义,后来由于语法原因,该定义在 2014 年进行了修订,即“当给予足够数量的时候,能够赋予宿主健康益处的一种活的微生物。” (FAO/WHO, 2002; Hill et al., 2014).
Hill 等人(2014)规定益生菌必须“有明确的含量,在货架期结束前有适当的活性计数和对健康有益的适当证据”,并进一步指出,所有益生菌必须保证“对其预期使用是安全的”。2018 年,国际益生菌和益生元科学协会(ISAPP)在一份立场声明中重申了这些观点。
即便有明确的定义,“益生菌”一词在食品和补充剂类别中仍被广泛使用,且经常被误用。在这里,我们提供了正确使用益生菌术语所需的最低标准。当新的“生物”术语被引入全球通用语言时(例如药物学、心理生物学、后生物学、合生学等术语),这一点尤其有用。
益生菌定义中的每一部分内容都很重要,并都可以被“转换”为易于使用的标准。定义这些标准一直是益生菌领域不同利益相关者的主要目标。
无论最终应用如何,这些标准可以表示为如图 1 所示的决策树,以确定候选菌株或菌株组合是否符合益生菌标准。此外,它们还可以以清单(checklist)的方式呈现,例如 ISAPP 信息图(ISAPP,2019),或者按照 Toscano 等人建议的以“条例(commandments)”列表的形式呈现(2017)。
本文所包含的益生菌的定义并不局限于传统的益生菌。当然,创新将使候选益生菌从新的来源中分离出来,这些新的来源具有当前无法预期的功能以及令人振奋的新健康益处。此定义不排除这些所谓的下一代益生菌,并且在某些情况下可能被概念化为活体生物药。
但是,根据预期用途,在开发此类益生菌时必须合理解决安全、法律和伦理问题,例如在适用的情况下遵守《名古屋议定书》(Nagoya protocol)(Johansen,2017 年),以及在将微生物从人类分离的情况下确保一定的知情同意。
尽管有较早的出版物,但仍需要明确细致地规定这些标准,并提供有关实现这些标准的详细信息(不涉及潜在的机械要求的细节)。 简而言之,益生菌菌株必须(i)充分被表征;(ii)预期用途安全;(iii)至少有一项根据公认的科学标准进行的人类临床试验,并获得阳性结果;(iv)在产品的整个货架期内保持有效剂量。
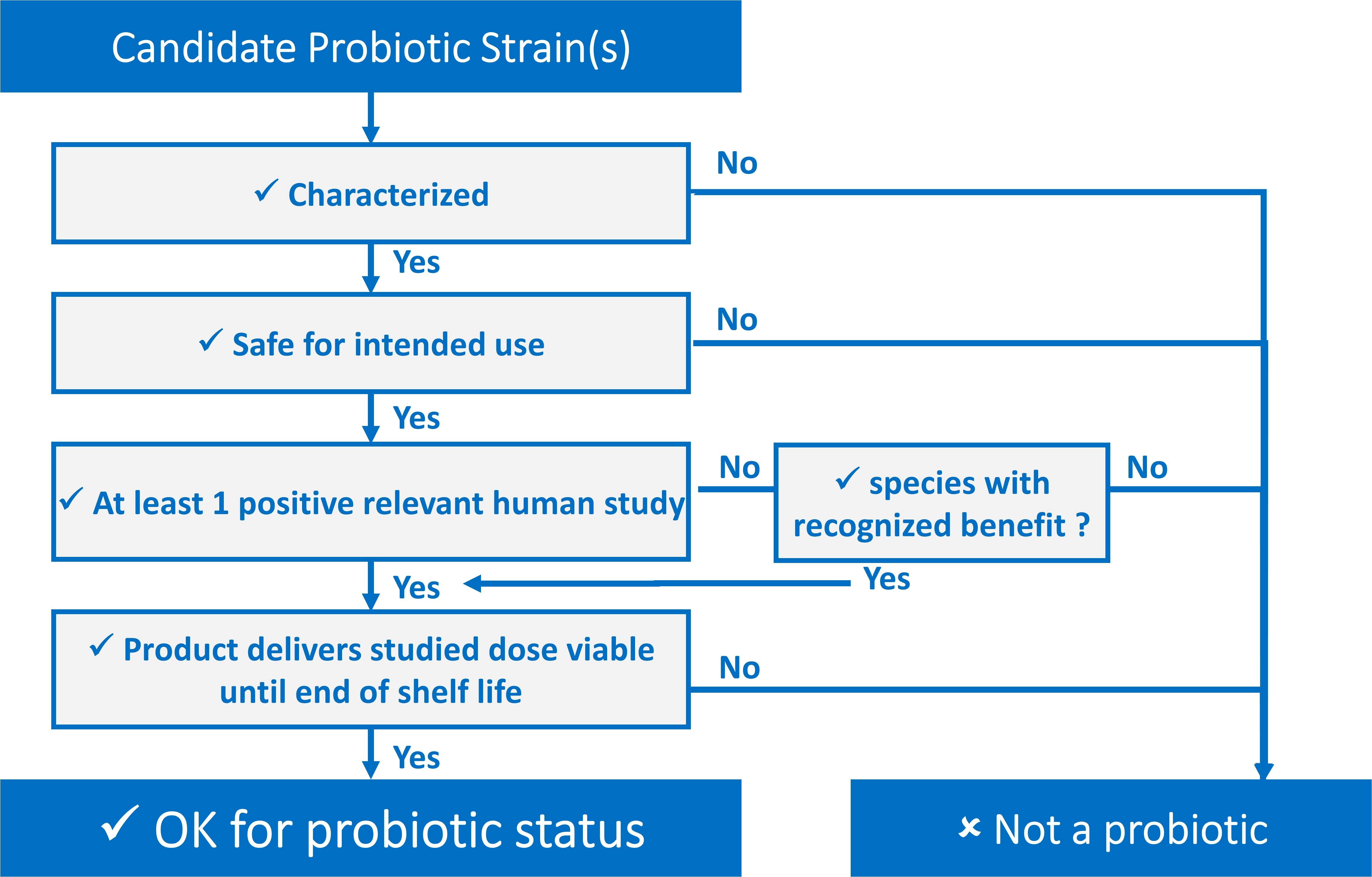
图1.确定候选益生菌是否符合标准的决策树
正确表征益生菌的关键是正确的菌株鉴定和命名。益生菌菌株应根据当前有效的细菌命名法——国际命名法(Parker 等人,2019)命名。最新的原核生物名称和命名法可在http://www.bacterio.net/ 上获得(Parte,2018)。
益生菌的鉴定需要确定菌株所属的已建立的并命名的属和种,以及亚种。由于某些益生菌活性可能是菌株特异性的,因此需要进一步正确分类菌株。
正确的菌株名称由两个主要部分组成:根据命名规则命名的官方属、物种(和亚种)名称,以及菌株名称。菌株名称可以是公认的菌种中心保藏目录号或商业菌株名称。
为此,我们建议将菌株保存在公认的菌种保藏中心中,以便安全保存,以及便于该菌株可用于研究,但不一定用于商业用途。
另外,为避免造成混淆,应避免对单个菌株使用多个菌株名称。制造商还应确保保持其菌株的遗传纯度,以使产品随时间推移仍包含具有相同特性的相同菌株。
鉴定技术可能会因生物体的不同而有所差异,并且会随着时间的推移而发展,但是由于表型技术不足以进行正确的识别,因此一些分子方法目前已被广泛应用。
比如,使用可靠的参考序列,对 16S rDNA 的测序是一种众所周知的可靠的鉴定物种的方法。首先可以将获得的序列与覆盖几乎整个已知细菌多样性的大型参考数据库进行匹配,但最好使用诸如 PATRIC 之类的精选数据库进行最终验证(Wattam 等,2017)。
如有疑问或需要更详细的鉴定时,比如涉及其他分子和表型特征,可参考 Bergey’s Manual of Determinative Bacteriology。
此外,Mattarelli 等(2014)阐述了“描述双歧杆菌属、乳酸杆菌属和相关属的新分类群的推荐最低标准。”这对于正确鉴定而言,也是十分有用的资源。
菌株鉴定的“黄金标准”是包括染色体外元件(extrachromosomal elements)在内的全基因组测序(WGS)。完整的基因组允许鉴定微生物的物种和菌株水平。
参考数据库是有帮助的,例如具有完整的基因组序列的 NCBI 具有许多优势,因为它允许进行可靠而精确的菌株特异性鉴定,并有助于查找是否存在危险因素。此外,这将有助于鉴定常见于某些乳酸菌中,并且可能对益生菌活性很重要的质粒。
虽然 WGS 是首选方法,但其他分型方法可能允许对不同菌株进行比较。多位点序列分型(MLST)或脉冲场凝胶电泳可以用于比较菌株,但不能从头开始鉴定物种或属。
鉴定最好由专门的或获得一定认证的实验室完成,这些实验室可以访问所需的参考数据库并使用当前经过验证和校准的方法和设备来完成鉴定工作。
还应强调的是,对于益生菌混合物,正确识别混合物中的每个菌株都很重要,尤其是当每个菌株都符合益生菌资格时。
益生菌菌株的特征应支持其益生功能。虽然益生菌功能的声称需要临床结果(详见后文),但对被认为对益生菌功效具有重要意义的特征测试或可表明所观察到的临床结果的潜在机制。
这些特征可能包括相关身体部位的存活情况,乳酸或其他短链脂肪酸的产生,粘液或肠上皮细胞的粘附特征,与人体免疫细胞的相互作用,对消化酶、胆汁或酸的抵抗力,通过竞争排斥或产生细菌素或过氧化氢的抗菌活性。
为此,比利时高级卫生委员会发布了一份关于益生菌表征方法的报告(Huys 等,2013),重点放在鉴定、菌株分型和安全性评估方面。
然而,必须强调的是,这种表型特征并不是益生菌的必要条件。这些检测可能提供功能的指征或在初始筛选策略中有用,但它们不是益生功能的有效生物标志物。具有上述任何或全部特征的菌株不能仅基于这些基因型或表型的存在而使用益生菌一词。
最近的荟萃分析证实,某些健康影响的现有证据与特定益生菌菌株密切相关(McFarland 和 Evans,2018 年;van den Akker 等人,2018 年)。
在某些情况下,健康益处可能并不局限于特定的菌株,而是可以存在于更广泛的分类学群中(Sanders 等人,2017 年)。该功能可能与益生菌微生物的单一特性有关。一个例子是乳糖酶调节乳糖不耐受症状。像这样的特征的存在可能是物种的,而不是菌株特异性的。
但是,即使是由共同特征介导的健康益处,也必须在菌株水平上鉴定益生菌。

为消费者提供符合食用安全标准的食品和膳食补充剂是益生菌生产商的基本责任。要确定一种具体的益生菌可安全用于食品和膳食补充剂,首先需要正确识别菌株水平,并通过历史证据或实验进一步证明安全性。
安全使用的历史数据可能是对预期使用的安全性进行总体评估的重要因素。
在缺乏安全性的情况下,必须根据科学原则确定安全性,包括进行充分的 1 期研究(Brodmann等人,2017年)。菌株的安全性是根据具体案例进行评估的,无法针对证据做出普适的要求,但是益生菌菌株需要符合国家/地区监管机构规定的安全要求,正如下所述。
益生菌品种和菌株必须安全供人类消费
对于普通健康人群日常使用的益生菌而言,必须解决由于施用活微生物而引起的潜在安全问题。
大多数市售益生菌菌株多属于乳酸菌、双歧杆菌和酵母,它们被认为可安全用于食品和补充剂。这是因为无论作为益生菌,还是作为发酵剂,它们属于具有安全使用历史记录的属和种。(Bourdichon 等人,2018)。
除了安全使用的历史之外,欧洲食品安全局(EFSA)自 2007 年以来一直根据“合格安全推定(QPS)”概念维护被推定为可供人类安全食用的菌种清单(EFSA,2007)。
QPS 方法是一种循证的、全面且定期更新的方法,用于评估特定微生物物种的安全性。该名单是确定属于 QPS 品种的食品菌株安全性的合理依据,但是前提是进行下面描述的菌株特异性检测。
该名单来自历史数据、对知识体系的定期监控以及广泛的科学文献回顾,这些结果适用于过去在食物链中发现的一系列微生物(EFSA,2020b)。
应当指出的是,QPS 的范围是普通健康人群的食品消费,并未特别考虑弱势人群的潜在风险(EFSA,2005)或非食品使用的益生菌。
此外,QPS 清单并不是详尽无遗的,因为它是基于向 EFSA 提交的用于批准在欧盟市场上市的信息确定的,所以传统发酵食品中使用的许多微生物并不在该清单中(Bourdichon 等人,2018)。
在欧洲,如果某个菌株不属于 QPS 物种,则可能要在其投放市场之前使用新食品法规(欧盟,2015 年)进行评估。其他司法管辖区还有其他程序来评估益生菌的安全性,例如美国公认的安全性(GRAS)法规。
益生菌菌种和菌株特异性安全标准
根据“介绍”部分所概述的原则,任何安全性评估均基于正确的物种鉴定。此外,还需要识别属或物种特定的危险因素,并在菌株水平进行检测。其中最重要的是不具有获得性抗菌素耐药基因或已知的毒力因子。
在欧盟内部,EFSA 已经发布了几项指南,描述了相关抗生素的表型敏感性阈值和耐药性值以及确定这些值的方法(EFSA,2018 年)。该指南还应用于评估酵母菌对细菌和抗真菌药物的敏感性(EFSA,2018)。
标准化的分析方法可用于候选细菌菌株的表型筛选(ISO-IDF,2010)。如果观察到抗性值高于阈值,则需要进一步表征。
菌株的 WGS 将确认是否存在与所观察到的抗性有关的已知基因。如果检测到推定的抗性基因,建议确定转座因子是否在其基因组附近。如果在附近,则不能排除抗性基因是可转移的,不建议将该菌株商业化。
此外,基因组序列可以通过搜索至少两个数据库来帮助鉴定假定的抗药性基因。对于数据库中代表性不足的微生物,建议使用隐马尔可夫模型(Hidden-Markov 模型)数据库。
根据分类和预期用途,可能需要评估该菌株的基因组中是否存在编码已知毒力因子(例如毒素、入侵和粘附因子)的基因(EFSA,2020a)。
在抗生素抗性阈值未知的情况下,生产者有责任确保所提议的益生菌菌株不包含可转移的抗生素耐药性基因,并且保证耐药性图谱与同一物种的其他成员一致。
在某些情况下,可能有必要针对所考虑的分类单元的敏感性概况生成新数据,包括确保敏感性测试方法与所考虑的微生物的生理适应、相关。
其他表型特性可以在菌株水平上进行安全性评估,例如形成生物胺和 D-乳酸的能力。两者都可以通过基因组分析或标准化表型测试便捷地进行检测。此外,有时可以在菌株水平上评估溶血活性和胆汁盐水解酶活性,但它们与安全性的相关性尚待确定(Huys 等人,2013 年)。
体内安全性测试
就目前属于 QPS 并且有在食品中安全使用的历史记录的大多数益生菌而言,体内安全性测试的价值尚不清楚,特别是考虑到欧盟的立场,即出于伦理和效率原因,不应在动物身上进行不必要的研究(EU,2010)。在健康的动物模型(例如小鼠或大鼠)中,预计 QPS 菌种几乎不会产生任何影响(Shokryazdan 等人,2016)。
另一方面,人类干预研究可通过批判性监控和报告的不良事件来正确记录益生菌的安全性和耐受性。可以监测包括生命体征在内的生物学和临床参数,以收集有价值的安全数据。与基线或标准值相比存在意外偏差表示可能存在安全隐患。
在任何类型的临床干预中都必须记录这些安全终点,并根据公认的人体研究科学标准进行分析和报告。赞助商、研究人员、作者和期刊编辑应促进系统地报告益生菌人类临床干预中的安全性和耐受性数据。
值得注意的是,任何研究,特别是持续时间较长、涉及大量受试者的研究,都肯定会观察到不良事件。关键是要确定干预组之间的不良事件是否不同(即益生菌和安慰剂)和/或是否被认为与干预相关。
到目前为止,在健康受试者的研究中只报告了罕见的、轻微的和短暂的益生菌相关不良事件(Goldenberg 等人,2017 年)。对于存在特定敏感的人群(例如年轻人、老年人、孕妇和免疫受损人群),建议对这些人群进行益生菌干预和医疗监督(Sanders 等人,2016 年)。
由于乳酸菌、双歧杆菌和酵母菌中的大量菌株具有长期的安全使用历史,并且已经过全面评估和监测,因此可以得出结论,对于普通人群来说,它们在食品和膳食补充剂中的使用没有重大的安全问题。食品和膳食补充剂安全性评估的重点是预期用途;其他用途可能有不同的安全要求,例如,取决于它们的投放形式或剂量。

自 2001 年以来(FAO/WHO,2002),赋予宿主健康益处的能力一直是益生菌定义的基本组成部分,并在 2014 年得到了重申(Hill 等,2014)。所谓健康益处,是指使用益生菌对人的健康产生一定的积极影响。此措辞是开放的,允许在探索可能的健康终点时进行创新。
但是,需要至少有一项证明对健康有益的人体试验才能使候选微生物菌株符合益生菌标准,并且最好在随后进行验证性试验。
在此,我们声明必须根据公认的科学标准进行试验。如某些权威机构所认可的那样,在极少数情况下,“益生菌”一词可能适用于属于同一菌种(或其他分类群)的多个菌株,因为其中的许多菌株拥有共同的机制来改善健康(Hill 等人,2014 年;Sanders 等人,2017 年)。
例如,嗜热链球菌或德氏乳杆菌亚种的菌株表达乳糖酶活性,而保加利亚乳杆菌可以减少与乳糖消化不良相关的症状,这是这些物种的共同特征。基于这种益处,嗜热链球菌和保加利亚乳杆菌菌株可以被认为是“益生菌”。
此外,为了正确地使用术语“益生菌”来描述此类菌株,必须在菌株水平对该生物进行鉴定,并证明其表现出相关特性。
对健康有益的有效证明取决于试验本身的质量和健全性(即设计和实施的良好程度)以及科学界对已发表的试验结果进行批判性评估的能力(即报告的良好程度)。现有几种工具可用于评估益生菌临床试验的设计、报告、偏倚风险(ROB)评估和关键评估(表 1)。
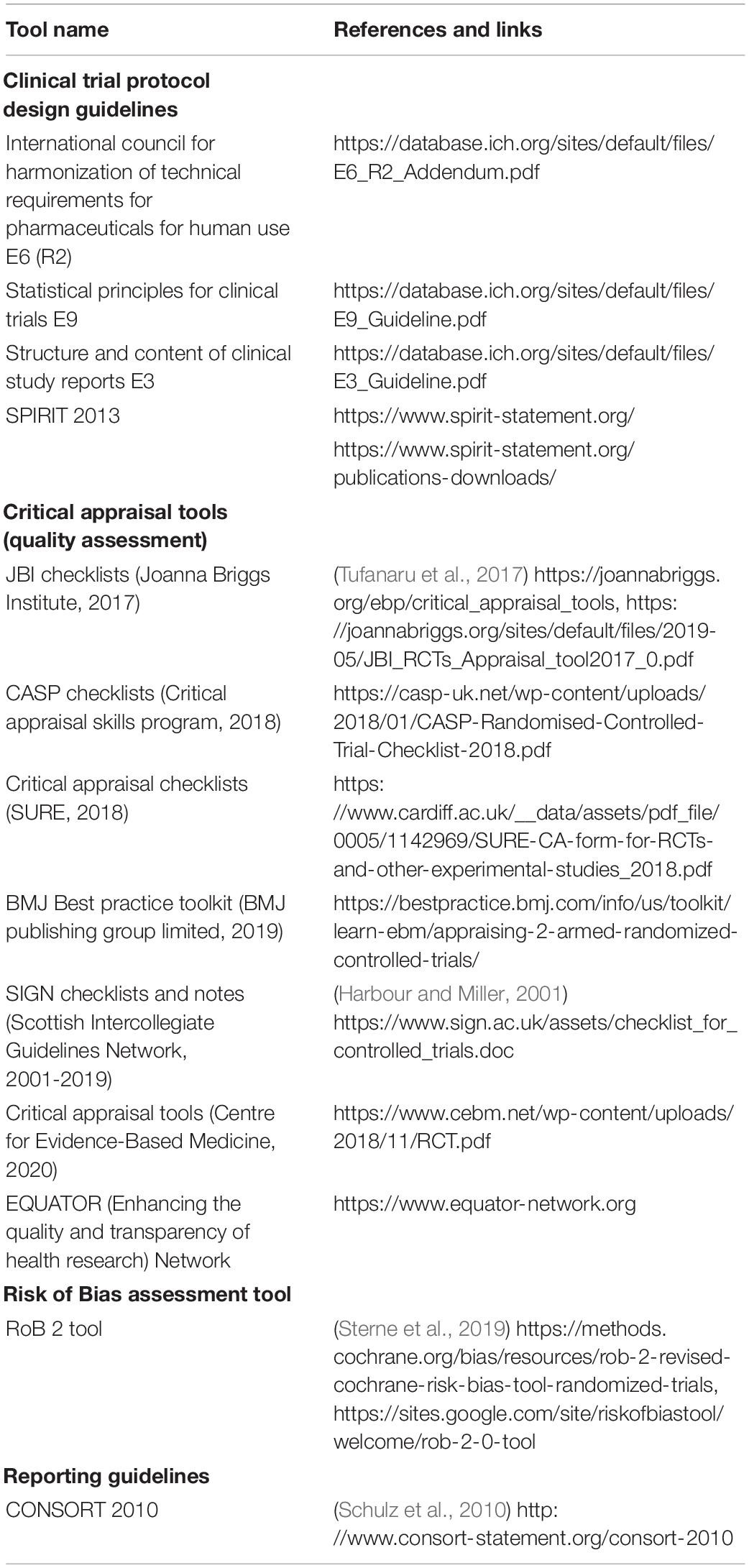
表1. 促进、报告、管理偏倚风险以及对益生菌人类干预研究的批判性报告的工具
方案设计的注意事项
最初制定公认的临床试验设计(和实施)指南是为了确保参与者的福利和符合伦理的临床试验行为,这一指南已经实行了几十年,而且往往是强制性的,例如 The Good Clinical Practice guidelines of the International Council for Harmonisation(ICH-GCP)以及不同国家/地区各自的具有法律约束力的规定(Vijayananthan 和 Nawawi,2008 年)。
遵从 ICH-GCP 指南,除了能够提供明确的伦理价值外,还有助于确保生成更高质量和更可靠的数据。这是一种国际公认的工具,得到了世界各地期刊、赞助者、监管机构和学术机构的认可,为符合国际医学杂志编辑委员会(ICMJE)建议的试验方案而专门开发的。
2013 年的“标准方案项目:介入试验建议(SPIRIT)”检查表包含了所有临床试验方案中应该包括的 33 种元素的清单(Chan 等人,2013 年)。根据 SPIRIT 2013 清单(第 2 项),强烈建议在研究开始之前在公共数据库(例如 ClinicalTrials.gov)中发布研究方案,这被视为促进更高质量研究设计的一种方式,同时有助于更透明地报告结果。
此外,在同行评审期刊上发表临床试验方案通常需要在公共登记处进行注册,这也是益生菌领域应进一步予以鼓励的良好做法。
从科学的角度来看,一些试验设计挑战似乎经常阻碍在益生菌试验中得出关于健康益处的正式结论(Brussow,2019)。
这些试验设计可能涉及研究设计的详细信息随机对照试验(RCT)与非随机试验,交叉对照试验与平行对照设计,参与者的分配方案隐藏,盲法(双盲与单盲或开放标签),对照的选择(安慰剂与对照治疗),剂量和给药方案(使用的浓度、给药程序、补充期的开始和持续时间),主要结果的功效计算或人群的选择(健康状况、年龄和性别)。
由于候选微生物菌株的固有特异性,目前还没有针对整个益生菌领域的指南来说明某一研究设计类型的偏好,或特定给药方案的偏好。但是,在研究设计阶段,有必要仔细考虑这些参数以及微生物菌株和目标群体的特征(Shane 等人,2010)。为此,事先了解公认的试验报告标准以及对已发表试验进行批判性评估的可用工具可能会有所帮助(表 1)。
试验报告的注意事项
一些国际期刊要求作者根据一套既定且公认的准则,即 Consolidated Standards of Reporting Trials (CONSORT),报告其试验结果,该准则已成为报告和发表试验结果的主要依据(Schulz 等,2010)。
虽然未将含有 25 点的 CONSORT 清单作为试验设计和实施的指南,但事先了解必须报告的因素可以帮助设计。在制定 SPIRIT 2013 协议设计指南时考虑了 CONSORT 2010 清单,以促进符合 SPIRIT 的协议与符合 CONSORT 报告的兼容性(Chan 等,2013)。
遵守 CONSORT 2010 试验报告指南将有助于对结果进行后续的批判性评估,并有助于通过荟萃分析和系统评价得出更强有力的结论。
简而言之,CONSORT 涵盖了试验设计和实施、数据收集和分析以及报告的方方面面。
例如,CONSORT 要求制作一份参与者流程图,明确说明从招募到研究完成的每个步骤的参与者人数、排除情况、随访损失以及意向性治疗或按方案治疗的人群的规模。这些信息对于以后的质量和 RoB 评估至关重要(例如,针对循证医学指南或监管目的进行系统评价)。
CONSORT 还规定,作者应强调其研究的局限性,例如可能影响结果解释的偏见和不确定因素。期望对结果进行概括性的描述,以及对健康益处与风险进行清晰的对比(意味着对不良事件进行详细报告)。
对已发表的试验进行批判性评价和 RoB 评价
临床研究的批判性评估和 RoB 评估是循证医学的重要组成部分。它们允许以客观的方式确定试验结果的权重(Buccheri 和 Sharifi,2017)。
为了这些目的,目前已经开发了许多工具,这些工具主要是为系统评价和荟萃分析或最佳实践指南的作者设计的,不过在评估未使用如 CONTORT 2010 报告指南的的情况下发表的试验的质量时可能会很有用。这主要涉及 2010 年之前发表的试验,但不幸的是,也涉及了部分较新的试验。总体上,在医学文献中,对 COSORT 2010 指南的遵从性仍然很低(Jin 等人,2018)。
批判性评估(即质量评估)工具和 RoB 评估工具之间的区别可能被认为是模棱两可的,但这两种方法显然是截然不同的。例如,Cochrane 综述的作者使用 RoB 评估工具旨在确定试验结果是否存在偏倚和可信度的问题(Higgins 等人,2019 年;Sterne 等人,2019 年)。
另一方面,质量评估工具通常包括与报告质量(例如,获得伦理批准或描述功效的计算)以及研究的质量、透明度和一致性(例如,随机化和分配方案隐藏、适当的对照选择和丢失的结果数据)有关的参数。后两个参数与 RoB 工具评估的潜在偏倚来源直接相关。
一般来说,试验结果的权重与避免偏倚来源的效率成正比(Higgins 等人,2019 年)。
考虑到临床试验中缺乏随机化、盲法或对照是产生偏倚的重要来源,双盲随机对照试验已成为以可靠的方式证明健康益处的“黄金标准”设计,正如随机对照试验在对临床试验证据质量进行分级时比其他设计获得更高的分数所证明的那样(Guyatt 等人,2008 年)。
其他研究设计,如开放标签和非对照研究,也是可以使用的试验设计,过去也曾使用过。虽然这类研究的结果可能不够可靠,不足以单独用于确定益生菌的资格,但它们可以提供有用的支持性数据。
虽然前面描述的三个标准专门针对被视为益生菌的微生物菌株,但第四条标准适用于提供益生菌的产品。
益生菌的定义没有提及特定的剂量,而是指出应以足以对宿主产生健康益处的量施用益生菌。
因此,可以想象的是,鉴于益生菌是能够在宿主内自我复制的活微生物,随着时间的推移,如果益生菌在宿主内生长充分,几个益生菌细胞就足以产生有益的效果。致病微生物当然也是如此,极低剂量的致病微生物也可能会对宿主健康造成有害影响,因为它们的毒力和在宿主内复制的能力。
剂量范围研究的目的是确定一种活性物质的耐受性、有效性和安全性,这种活性物质可以以固定浓度给药,而且通常不能在给药后倍增(Ting,2006)。因此,剂量范围研究是临床试验中的常见特征,但在食品和膳食补充剂试验中较少见。这主要是因为食品成分的安全性被假定。
在临床环境中,通常在阐明了所研究的生物活性物质的最大耐受剂量(MTD)之后,才进行剂量范围研究。我们尚不了解在人类中针对任何益生菌菌株或菌株组合进行的任何口服 MTD 研究。
鉴于益生菌具有极好的安全性,而且很少进行 MTD 或剂量范围研究,因此大多数研究通常选择每天在 108 至 1011 个菌落形成单位(CFU)之间的剂量,这反映了以往研究中的有效剂量。虽然人们可能对确定导致特定健康益处的最佳剂量感兴趣,但这不是一个必要的标准。
益生菌菌株活力的量化应使用标准化的计数方法,如平板法——选择性生长培养基上的 CFU 计数,例如嗜酸乳杆菌(ISO 20128)和双歧杆菌(ISO 29981 或 IDF 220:2010)或通过流式细胞仪(ISO 19344:2015;表 2)进行计数。这些方法由多位作者进行了汇总(Davis,2014;Zielińskaet al,2018)。
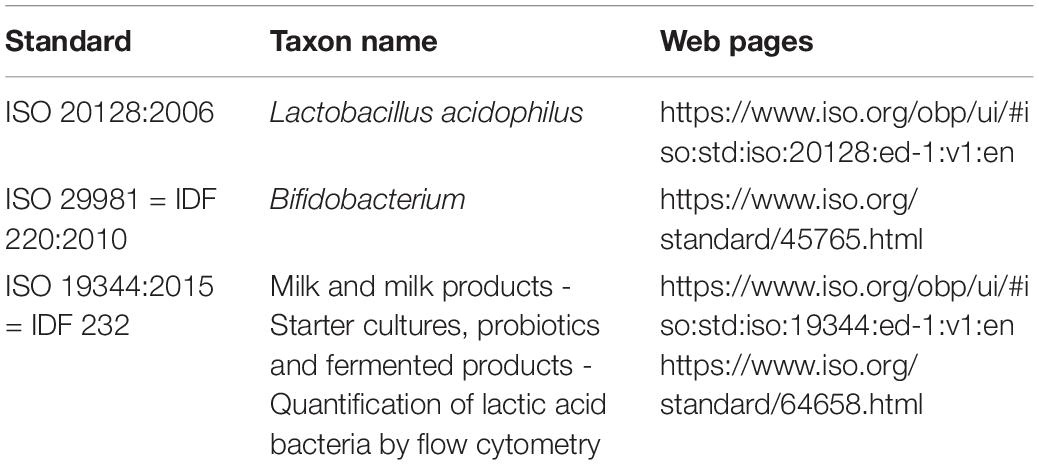
表2.益生菌计数标准方法示例
有效剂量下益生菌菌株的生存能力应在临床研究期间记录在测试产品中,并遵守质量程序,保证产品到保质期结束时都具有有效剂量。
为了量化益生菌组合,已经开发了基于高通量下一代测序的不依赖培养物的宏基因组学方法(Patro等人,2016),尽管这些方法不能确保所计数的微生物是有活性的。但这些方法可以揭示潜在污染物的信息(Quigley等,2013),但可能缺乏方法学的验证(Sohier 等,2014)。
然而,它们可以由获得认可的实验室进行,以保证一定程度的一致性和重现性。
目前正在开发确定复杂混合物中活性的分子方法,例如单叠氮丙啶(PMA)-PCR(Scariot等,2018)。 但是,这些只是试验性的,并不是标准化的。
粪便恢复通常被用作替代标记物,以反映胃肠道健康目标所需的足够剂量。对于那些容易确定功效终点(例如血清胆固醇水平)的益生菌来说,剂量范围可能是能确定的,尽管原位复制的问题再次可能导致解释上的问题。
一项涉及婴儿长双歧杆菌亚种 35624 的 IBS 试验进行了剂量范围研究,测试了 106、108 和 1010 CFU 三种剂量。结果很有趣,108 CFU 剂量是有效的,而其他两个剂量则无效。这种异常结果很可能是因为含有最高剂量的胶囊没有溶解,因此只能评估其他两个剂量的结果(Whorwell等,2006)。
经典剂量范围研究的另一种方法是检查大量已记录的人类益生菌试验,并计算每次研究中使用的剂量和临床结果。最近进行了这样的分析,并得出了微妙的结论(Ouwehand,2017)。对于某些益生菌/健康状态组合,有明显的剂量反应证据,但对于其他组合,数据并不令人信服。
如果某项研究得到了预期的健康益处,那么该剂量将作为健康声明应被允许的最低剂量。使用更高剂量的产品也能够提出同样的声明,但任何低于人体测试剂量的声明都不应该被允许。
在这里,我们不会进一步讨论健康声明,这属于监管机构的范畴。虽然监管机构总体上遵守益生菌的定义,但他们倾向于在各自的管辖范围内对要求作出不同的解释。对此的分析超出了本文的范围。
递送形式对益生菌作用有什么影响(如果有的话)是一个有趣的话题。迄今为止,很少有研究对采用同一临床终点,但使用不同基质递送益生菌的效果进行探究。
有两篇综述讨论了这一问题,一篇得出的结论是,目前没有证据表明递送基质对益生菌功效有实质性影响(Sanders 等,2014),另一篇综述得出的结论是,可能存在菌株依赖的基质效应(Flach等,2018)。两篇论文都认为有关该主题的数据很少。
益生菌是全球调查研究、创新产品设计、有效营销、监管审查、消费者兴趣和保健从业者关注的主题。清楚地理解并负责任地使用“益生菌”一词所需的标准,对所有参与这些工作的人都是有益的。
本文介绍了适用于食品和膳食补充剂中的益生菌菌株的最低标准,类似的标准也可能适用于益生菌的其他用途。
具体而言,该菌株必须使用公认的科学方法进行鉴定,根据当前有效的命名法命名,并使用可检索的菌株名称命名。方法将取决于益生菌的种类,并且可能会随着技术的发展而改变。此外,我们还建议将其存放在国际菌种保藏库中。
此外,该菌株必须已证明其预期用途的安全性,并至少拥有基于一项符合公认的科学标准或符合当地/国家主管部门的建议和规定(如果适用)的研究,证明其具有健康益处。
在整个保质期内,产品都必须包含足够量的益生菌菌株,以便能够提供所声称的(和基于证据的)健康益处。
产品应根据适用的良好生产要求进行生产,以确保安全性、纯度和稳定性(Jackson 等,2019),并且产品标签应该能够向终端用户传达产品的基本信息(具体菌株、保质期结束时的活益生菌水平以及允许的健康益处声明)。
遵守这些原则将确保市场上没有滥用“益生菌”一词的产品。一些地方监管机构可以用不同的方式定义益生菌,但产品制造商有责任生产和销售符合当地规章制度并符合上述原则的益生菌。
参考文献:
1.Bourdichon,F.,Alper,I.,Bibiloni,R.,Dubois,A.,Laulund,S.,Miks,M.,etal.(2018).Inventoryofmicrobialfoodcultureswithsafetydemonstrationinfermentedfoodproducts.Bull.Int.DairyFederat.495,5–71.
2.Brodmann,T.,Endo,A.,Gueimonde,M.,Vinderola,G.,Kneifel,W.,deVos,W.M.,etal.(2017).Safetyofnovelmicrobesforhumanconsumption:practicalexamplesofassessmentintheEuropeanUnion.Front.Microbiol.8:1725.doi:10.3389/fmicb.2017.01725
3.Brussow,H.(2019).Probioticsandprebioticsinclinicaltests:anupdate.F1000Res8:F1000FacultyRev-1157.doi:10.12688/f1000research.19043.1
4.Buccheri,R.K.,andSharifi,C.(2017).CriticalAppraisalToolsandReportingGuidelinesforEvidence-BasedPractice.WorldviewsEvid.BasedNurs.14,463–472.doi:10.1111/wvn.12258
5.Chan,A.W.,Tetzlaff,J.M.,Altman,D.G.,Laupacis,A.,Gotzsche,P.C.,Krleza-Jeric,K.,etal.(2013).SPIRIT2013statement:definingstandardprotocolitemsforclinicaltrials.Ann.Intern.Med.158,200–207.doi:10.7326/0003-4819-158-3-201302050-201302583
6.Davis,C.(2014).Enumerationofprobioticstrains:reviewofculture-dependentandalternativetechniquestoquantifyviablebacteria.J.Microbiol.Methods103,9–17.doi:10.1016/j.mimet.2014.04.012
7.EFSA(2005).OpinionoftheScientificCommitteeonarequestfromEFSArelatedtoagenericapproachtothesafetyassessmentbyEFSAofmicroorganismsusedinfood/feedandtheproductionoffood/feedadditives.EFSAJ.3:226.doi:10.2903/j.efsa.2005.226
8.EFSA(2007).IntroductionofaQualifiedPresumptionofSafety(QPS)approachforassessmentofselectedmicroorganismsreferredtoEFSA-OpinionoftheScientificCommittee.EFSAJ.5:587.doi:10.2903/j.efsa.2007.587
9.EFSA(2018).Guidanceonthecharacterisationofmicroorganismsusedasfeedadditivesorasproductionorganisms.EFSAJ.16:5206.doi:10.2903/j.efsa.2018.5206
10.EFSA(2020a).EFSAStatementontheRequirementsforWholeGenomeSequenceAnalysisofMicroorganismsIntentionallyUsedintheFoodChain.Parma:EFSA.
11.EFSA(2020b).ScientificOpinionontheupdateofthelistofQPSrecommendedbiologicalagentsintentionallyaddedtofoodorfeedasnotifiedtoEFSA(2017–2019).EFSAJ.18:5965.doi:10.2903/j.efsa.2020.5965
12.EU(2010).OntheProtectionOfAnimalsUsedforScientificPurposes,ed.E.Parliament,(Brussels:EuropeanParliament).
13.EU(2015).Onnovelfoods,amendingRegulation(EU)No1169/2011oftheEuropeanParliamentandoftheCouncilandrepealingRegulation(EC)No258/97oftheEuropeanParliamentandoftheCouncilandCommissionRegulation(EC)No1852/2001,ed.E.Parliament,(Brussels:EU).
14.FAO/WHO(2002).GuidelinesfortheEvaluationOfProbioticsinFood.Paris:FAO,1–11.
15.Flach,J.,vanderWaal,M.B.,vandenNieuwboer,M.,Claassen,E.,andLarsen,O.F.A.(2018).Theunderexposedroleoffoodmatricesinprobioticproducts:reviewingtherelationshipbetweencarriermatricesandproductparameters.Crit.Rev.FoodSci.Nutr.58,2570–2584.doi:10.1080/10408398.2017.1334624
16.Goldenberg,J.Z.,Yap,C.,Lytvyn,L.,Lo,C.K.,Beardsley,J.,Mertz,D.,etal.(2017).ProbioticsforthepreventionofClostridiumdifficile-associateddiarrheainadultsandchildren.CochraneDatabase.Syst.Rev.12:CD006095.doi:10.1002/14651858.CD006095.pub4
17.Guyatt,G.H.,Oxman,A.D.,Vist,G.E.,Kunz,R.,Falck-Ytter,Y.,Alonso-Coello,P.,etal.(2008).GRADE:anemergingconsensusonratingqualityofevidenceandstrengthofrecommendations.BMJ336,924–926.doi:10.1136/bmj.39489.470347.AD
18.Harbour,R.,andMiller,J.(2001).Anewsystemforgradingrecommendationsinevidencebasedguidelines.BMJ323,334–336.doi:10.1136/bmj.323.7308.334
19.Higgins,J.P.T.,Thomas,J.,Chandler,J.,Cumptson,M.,Li,T.,Page,M.J.,etal.(2019).CochraneHandbookforSystematicReviewsofInterventions.London:Cochrane.
20.Hill,C.,Guarner,F.,Reid,G.,Gibson,G.R.,Merenstein,D.J.,Pot,B.,etal.(2014).Expertconsensusdocument.TheInternationalScientificAssociationforProbioticsandPrebioticsconsensusstatementonthescopeandappropriateuseofthetermprobiotic.Nat.Rev.Gastroenterol.Hepatol.11,506–514.doi:10.1038/nrgastro.2014.66
21.Huys,G.,Botteldoorn,N.,Delvigne,F.,DeVuyst,L.,Heyndrickx,M.,Pot,B.,etal.(2013).Microbialcharacterizationofprobiotics–advisoryreportoftheWorkingGroup“8651Probiotics”oftheBelgianSuperiorHealthCouncil(SHC).Mol.Nutr.FoodRes.57,1479–1504.doi:10.1002/mnfr.201300065
22.ISAPP(2018).MinimumCriteriaforProbiotics[Online].Sacramento,CA:InternationalScientificAssociationforProbioticsandPrebiotics.
23.ISAPP(2019).ProbioticChecklist:MakingaSmartSelection[Online].Sacramento,CA:InternationalScientificAssociationofProbioticsandPrebiotics.
24.ISO-IDF(2010).MilkandMilkProducts—DeterminationoftheMinimalInhibitoryConcentration(MIC)ofAntibioticsApplicabletoBifidobacteriaandNon-EnterococcalLacticAcidBacteria(LAB).London:ISO.
25.Jackson,S.A.,Schoeni,J.L.,Vegge,C.,Pane,M.,Stahl,B.,Bradley,M.,etal.(2019).ImprovingEnd-UserTrustintheQualityofCommercialProbioticProducts.Front.Microbiol.10:739.doi:10.3389/fmicb.2019.00739
26.Jin,Y.,Sanger,N.,Shams,I.,Luo,C.,Shahid,H.,Li,G.,etal.(2018).Doesthemedicalliteratureremaininadequatelydescribeddespitehavingreportingguidelinesfor21years?-Asystematicreviewofreviews:anupdate.J.Multidiscip.Healthc.11,495–510.doi:10.2147/JMDH.S155103
27.Johansen,E.(2017).Futureaccessandimprovementofindustriallacticacidbacteriacultures.Microb.CellFact.16:230.doi:10.1186/s12934-017-0851-851
28.Mattarelli,P.,Holzapfel,W.,Franz,C.M.,Endo,A.,Felis,G.E.,Hammes,W.,etal.(2014).RecommendedminimalstandardsfordescriptionofnewtaxaofthegeneraBifidobacterium,Lactobacillusandrelatedgenera.Int.J.Syst.Evol.Microbiol.64(Pt4),1434–1451.doi:10.1099/ijs.0.060046-60040
29.McFarland,L.V.,andEvans,C.T.(2018).Goldsteinstrain-specificityanddisease-specificityofprobioticefficacy:Asystematicreviewandmeta-analysis.EJC.Front.Med.(Lausanne)5:124.doi:10.3389/fmed.2018.00124
30.Ouwehand,A.C.(2017).Areviewofdose-responsesofprobioticsinhumanstudies.Benef.Microbes8,143–151.doi:10.3920/BM2016.0140
31.Parker,C.T.,Tindall,B.J.,andGarrity,G.M.(2019).InternationalCodeofNomenclatureofProkaryotes.Int.J.Syst.Evol.Microbiol.69,S1–S111.
32.Parte,A.C.(2018).LPSN-ListofProkaryoticnameswithStandinginNomenclature(bacterio.net),20yearson.Int.J.Syst.Evol.Microbiol.68,1825–1829.doi:10.1099/ijsem.0.002786
33.Patro,J.N.,Ramachandran,P.,Barnaba,T.,Mammel,M.K.,Lewis,J.L.,andElkins,C.A.(2016).Culture-IndependentMetagenomicSurveillanceofCommerciallyAvailableProbioticswithHigh-ThroughputNext-GenerationSequencing.mSphere1:e00057-16.doi:10.1128/mSphere.00057-16
34.Quigley,L.,McCarthy,R.,O’Sullivan,O.,Beresford,T.P.,Fitzgerald,G.F.,Ross,R.P.,etal.(2013).Themicrobialcontentofrawandpasteurizedcowmilkasdeterminedbymolecularapproaches.J.DairySci.96,4928–4937.doi:10.3168/jds.2013-6688
35.Sanders,M.E.,Benson,A.,Lebeer,S.,Merenstein,D.J.,andKlaenhammer,T.R.(2017).Sharedmechanismsamongprobiotictaxa:implicationsforgeneralprobioticclaims.Curr.Opin.Biotechnol.49,207–216.doi:10.1016/j.copbio.2017.09.007
36.Sanders,M.E.,Klaenhammer,T.R.,Ouwehand,A.C.,Pot,B.,Johansen,E.,Heimbach,J.T.,etal.(2014).Effectsofgenetic,processing,orproductformulationchangesonefficacyandsafetyofprobiotics.Ann.N.Y.Acad.Sci.1309,1–18.doi:10.1111/nyas.12363
37.Sanders,M.E.,Merenstein,D.J.,Ouwehand,A.C.,Reid,G.,Salminen,S.,Cabana,M.D.,etal.(2016).Probioticuseinat-riskpopulations.J.Am.Pharm.Assoc.56,680–686.doi:10.1016/j.japh.2016.07.001
38.Scariot,M.C.,Venturelli,G.L.,Prudencio,E.S.,andArisi,A.C.M.(2018).QuantificationofLactobacillusparacaseiviablecellsinprobioticyoghurtbypropidiummonoazidecombinedwithquantitativePCR.Int.J.FoodMicrobiol.264,1–7.doi:10.1016/j.ijfoodmicro.2017.10.021
39.Schulz,K.F.,Altman,D.G.,Moher,D.,andGroup,C.(2010).CONSORT2010statement:updatedguidelinesforreportingparallelgrouprandomizedtrials.Ann.Intern.Med.152,726–732.doi:10.7326/0003-4819-152-11-201006010-201006232
40.Shane,A.L.,Cabana,M.D.,Vidry,S.,Merenstein,D.,Hummelen,R.,Ellis,C.L.,etal.(2010).Guidetodesigning,conducting,publishingandcommunicatingresultsofclinicalstudiesinvolvingprobioticapplicationsinhumanparticipants.GutMicrobes1,243–253.doi:10.4161/gmic.1.4.12707
41.Shokryazdan,P.,FaselehJahromi,M.,Liang,J.B.,Kalavathy,R.,Sieo,C.C.,andHo,Y.W.(2016).Safetyassessmentoftwonewlactobacillusstrainsasprobioticforhumanusingaratmodel.PLoSOne11:e0159851.doi:10.1371/journal.pone.0159851
42.Sohier,D.,Pavan,S.,Riou,A.,Combrisson,J.,andPostollec,F.(2014).Evolutionofmicrobiologicalanalyticalmethodsfordairyindustryneeds.Front.Microbiol.5:16.doi:10.3389/fmicb.2014.00016
43.Sterne,J.A.C.,Savovic,J.,Page,M.J.,Elbers,R.G.,Blencowe,N.S.,Boutron,I.,etal.(2019).RoB2:arevisedtoolforassessingriskofbiasinrandomisedtrials.BMJ366:l4898.doi:10.1136/bmj.l4898
44.Ting,N.(2006).DoseFindinginDrugDevelopment.NewYork,NY:Springer.
45.Toscano,M.,DeGrandi,R.,Pastorelli,L.,Vecchi,M.,andDrago,L.(2017).Aconsumer’sguideforprobiotics:10goldenrulesforacorrectuse.Dig.LiverDis.49,1177–1184.doi:10.1016/j.dld.2017.07.011
46.Tufanaru,C.,Munn,Z.,Aromataris,E.,Campbell,J.,andHopp,L.(2017).“Systematicreviewsofeffectiveness,”inJoannaBriggsInstituteReviewer’sManual,edsE.Aromataris,andZ.Munn,(Adelaide:JoannaBriggsInstitute).
47.vandenAkker,C.H.P.,vanGoudoever,J.B.,Szajewska,H.,Embleton,N.D.,Hojsak,I.,Reid,D.,etal.(2018).Espghanworkinggroupforprobiotics,prebiotics&committeeonnutrition.probioticsforpreterminfants:Astrain-specificsystematicreviewandnetworkmeta-analysis.J.Pediatr.Gastroenterol.Nutr.67,103–122,doi:10.1097/MPG.0000000000001897
48.Vijayananthan,A.,andNawawi,O.(2008).TheimportanceofGoodClinicalPracticeguidelinesanditsroleinclinicaltrials.Biomed.ImagingInterv.J.4:e5.doi:10.2349/biij.4.1.e5
49.Wattam,A.R.,Davis,J.J.,Assaf,R.,Boisvert,S.,Brettin,T.,Bun,C.,etal.(2017).ImprovementstoPATRIC,theall-bacterialBioinformaticsDatabaseandAnalysisResourceCenter.NucleicAcidsRes.45,D535–D542.doi:10.1093/nar/gkw1017
50.Whorwell,P.J.,Altringer,L.,Morel,J.,Bond,Y.,Charbonneau,D.,O’Mahony,L.,etal.(2006).EfficacyofanencapsulatedprobioticBifidobacteriuminfantis35624inwomenwithirritablebowelsyndrome.Am.J.Gastroenterol.101,1581–1590.doi:10.1111/j.1572-0241.2006.00734.x
51.Zielińska,D.,Ołdak,A.,Rzepkowska,A.,andZieliński,K.(2018).“Enumerationandidentificationofprobioticbacteriainfoodmatrices,”inAdvancesinBiotechnologyforFoodIndustry,edsA.M.Holban,andA.M.Grumezescu,(London:AcademicPress),167–196.doi:10.1016/b978-0-12-811443-8.00006-2
原文链接:https://www.frontiersin.org/articles/10.3389/fmicb.2020.01662/full
作者|Sylvie Binda(达能), Colin Hill, Eric Johansen(科汉森), David Obis(达能), Bruno Pot(养乐多), Mary Ellen Sanders(ISAPP的执行科学官), Annie Tremblay (Rosell微生物和益生菌研究所)和 Arthur C. Ouwehand(杜邦营养与生物科学公司)
编译|Orchid
审校|617
编辑|笑咲